Inhaltsverzeichnis
Botanik Band II: Physiologie
Kapitel 2: Signalaufnahme und Transduktion
Eine Vielzahl interner Signale sind notwendig für die Koordination der Expression von Genen während der Entwicklung ebenso wie für die Fähigkeit der Pflanze, auf Umweltreize zu reagieren. Solche internen (ebenso wie externen) Signalstoffe bewirken ihre Effekte typischerweise durch biochemische Reaktionsketten: Signaltransduktionswege, die das ursprüngliche Signal hochgradig verstärken und letztlich zur Aktivierung oder Hemmung von Genen führen. [Taiz, 2002Taiz, Lincoln; Zeiger, Eduardo (2002): Plant Physiology, Sinauer Associates, Sunderland]
Der Signalaufnahme kommt in der Pflanze eine besondere Bedeutung durch ihre sessile Lebensweise zu. Da sie sich in aller Regel nicht wegbewegen kann, muß sie sich mit ihrer Umwelt arrangieren. Dazu gehört ein umfangreiches System zur Signalaufnahme und -verarbeitung, um angemessen auf Veränderungen der Umwelt reagieren zu können.
Lange Zeit waren die Signaltransduktionswege in planta nur sehr oberflächlich bekannt. Das hat sich im letzten Jahrzehnt langsam gewandelt, und mittlerweile sind z.B. fast alle Komponenten des Ethylen-Signalweges bekannt. Trotzdem sind wir noch weit von einem Verständnis der Zusammenhänge entfernt, da ihre Komplexität (cross-talking) gerade bei den Pflanzen immens ist.
Download
- Vorläufige Endfassung des zweiten Kapitels (PS.GZ, 662 KB) - Stand: 2003-11-28
Links
- The Protein Kinase Resource
The Protein Kinase Resource (PKR) aims to become a web accessible compendium of information on the protein kinase family of enzymes. This resource will include tools for structural and computational analyses as well as links to related information maintained by others. The PKR is a collaborative project of protein kinase researchers and computational biologists working to create a database integrating molecular and cellular information. - Taiz/Zeiger: Plant Physiology ONLINE - Chapter 14: Gene Expression and Signal Transduction
Chapter 14 is available in its entirety as an Adobe Acrobat (PDF) file.
Leider ist dieses Kapitel aus der 2. Auflage (1998) übernommen und stammt noch aus der Zeit vor der kompletten Sequenzierung des Arabidopsis-Genoms (2000). In der kurzen Zeit seitdem hat sich einiges auf dem Gebiet der Signaltransduktion getan. (Das Kapitel im Skript ist aktueller.)
Abbildungen
Bei den im folgenden vorgestellten „Web-Abbildungen“ handelt es sich um vom Autor für das Skriptum erstellte Abbildungen. Sie sollen einen ersten Eindruck von der Ausgestaltung des Kapitels vermitteln. Das Copyright aller Abbildungen liegt beim Autor.
Quellen
Die bei den Web-Abbildungen in eckigen Kammern angegebenen Quellen werden an anderer Stelle noch detailliert aufgeschlüsselt werden.
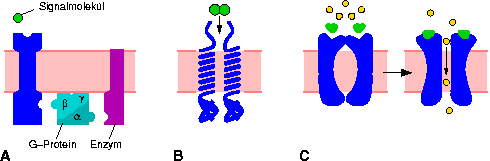
Web-Abb. 2.1
Membrangebundene Rezeptoren lassen sich in drei Klassen einteilen. (A) G protein-linked receptors; (B) enzyme-linked receptors; (C) ion channel-linked receptors. Stark schematisierte Darstellung. Nach [Buchanan, 2000Buchanan, B.; Gruissem, W.; Jones, R.L. (Hg.) (2000): Biochemistry and Molecular Biology of Plants, American Society of Plant Physiologists, Rockville], verändert.
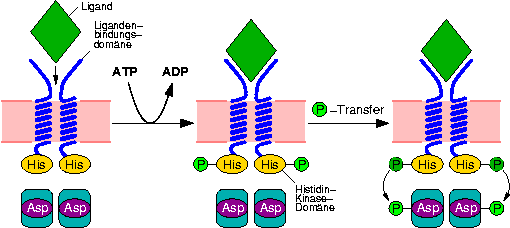
Web-Abb. 2.2
Die Rezeptoren (His-Kinasen) der Zwei-Komponenten-Systeme agieren als Homodimere. Bindet ein Ligand an den Rezeptor, wird dieser aktiviert und die beiden Monomere phosphorylieren einen bestimmten His-Rest am jeweils anderen Monomer. Anschließend wird der Phosphatrest auf einen konservierten Aspartrest eines response regulator Proteins übertragen. Dieser response regulator trägt häufig eine DNA-bindende Domäne und agiert als Transkriptionsfaktor. Bei den (in eukaryotischen Organismen ausschließlich vorkommenden) Hybrid-Kinasen ist die His-Kinase mit einer response regulator-Domäne verschmolzen. Ein weiterer response regulator ist typischerweise notwendig, um das Signal weiterzugeben. Nach [Buchanan, 2000Buchanan, B.; Gruissem, W.; Jones, R.L. (Hg.) (2000): Biochemistry and Molecular Biology of Plants, American Society of Plant Physiologists, Rockville], verändert.
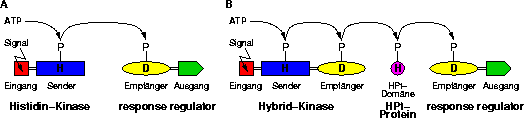
Web-Abb. 2.3
Schematische Darstellung und grundlegende Eigenschaften der simple (A) und der multistep (B) two-component signaling systems. Signalperzeption durch die Eingangsdomäne (input domain) der His- oder Hybrid-Kinase induziert Autophosphorylierung des Transmitters an einem konservierten His-Rest (H). Das Phosphat (P) wird an einen konservierten Asp-Rest (D) weitergegeben, der entweder auf dem Empfänger (receiver) des verwandten response regulator lokalisiert ist (A) oder, im Fall der Hypbrid-Kinasen, auf einer angehängen Empfänger-Domäne (receiver domain) (B). Beim multistep, two-component signaling system dienen His-tragende Phosphotransfer-Domänen-Proteine (Hiscontaining phosphotransfer (HPt) domain proteins) als Phospho-His-Intermediate zwischen den Hybrid-Kinasen und dem response regulator. Aus [Lohrmann, 2002Lohrmann, J; Harter, K (2002): Plant Two-Component Signaling Systems and the Role of Response Regulators, Plant Physiol. 128:363-369], verändert.
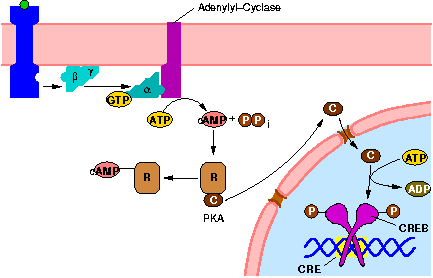
Web-Abb. 2.4
Die cAMP-vermittelte Signaltransduktion im tierischen Organismus: Ein Modell auch für Pflanzen? Die Adenylyl-Cyclase (AC) wird durch ein heterotrimeres G-Protein aktiviert und synthetisiert cAMP aus ATP. Das cAMP bindet an die regulatorische Untereinheit (R) der Proteinkinase A (PKA), die daraufhin dissoziiert und die katalytische Untereinheit der PKA freigibt. Die katalytische Untereinheit (C) der PKA wandert in den Zellkern und phosphoryliert dort den Transkriptionsfaktor CREB (cAMP response element-bindung protein), der daraufhin an die CRE-Sequenz (cAMP response element) im Promoter-Bereich cAMP-regulierter Gene bindet. Nach [Buchanan, 2000Buchanan, B.; Gruissem, W.; Jones, R.L. (Hg.) (2000): Biochemistry and Molecular Biology of Plants, American Society of Plant Physiologists, Rockville], verändert.
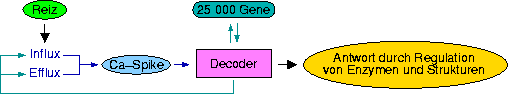
Web-Abb. 2.5
Allgemeine Übersicht über die Ca2+-Signaltransduktion. Codierung von Calcium-Signalen erfolgt über die Regulation von Influx und Efflux von Ca2+. Das führt zu einem definierten Ca2+-Spike. Decodierung von Calcium-Signalen führt zu spezifischen Antworten auf zellulärer Ebene. Verschiedene Rückkoppelungs-Mechanismen des Calcium-Sensors („Decoders“) sind möglich, z.B. die Regulation von Calcium-Spikes über die Kontrolle von Calcium-permeablen Kanälen oder über die Kontrolle der Pump-Aktivität. Aus [Sanders, 2002Sanders, Dale; Pelloux, Jerome; Brownlee, Colin; Harper, Jeffrey F. (2002): Calcium at the Crossroads of Signaling, Plant Cell 14:S401-417], verändert.
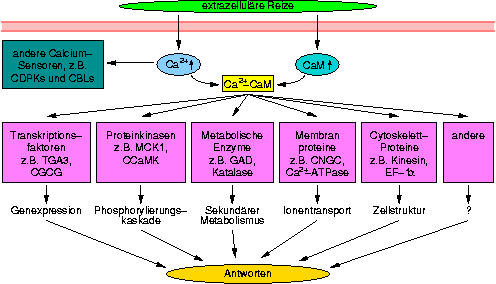
Web-Abb. 2.6
Modell des Ca2+/Calmodulin-vermittelten Signal-Netzwerkes in Pflanzen. Veränderungen des Ca2+-Signals werden durch Entwicklungs- oder Umweltsignale oder durch Hormone verursacht. Diese „Calcium-Signaturen“ werden durch Ca2+-Sensoren wie Calmodulin (CaM), Calcium-abhängige Protein-Kinasen (CDPK) und Calcineurin B-ähnliche Proteine (CBL) decodiert. Außerdem wird die Expression einiger CaM-Gene ebenfalls durch diese Signale induziert. Der aktivierte Ca2+/CaM-Komplex bindet an eine Vielzahl von Zielproteinen und verändert deren Aktivität. Zu diesen Zielproteinen gehören Transkriptionsfaktoren ebenso wie Proteinkinasen, metabolische Enzyme, Ionenkanäle und Transporter sowie Proteine des Cytoskeletts. Im Endeffekt führt das Ca2+/CaM-vermittelte Signal-Netzwerk zu physiologischen Reaktionen wie Zellwachstum oder Differenzierung, Streß-Toleranz oder Wachstumshemmung und Zelltod. Aus [Yang, 2003Yang, Tianbao; Poovaiah, B.W. (2003): Calcium/calmodulin-mediated signal network in plants, Trends in Plant Science 8:505-512], verändert.
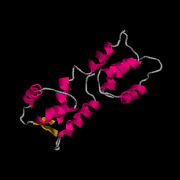
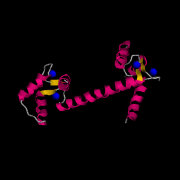
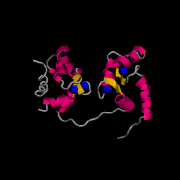
Web-Abb. 2.7
Strukturen von Calmodulin. (A) Apo-CaM (B) Ca2+-CaM (C) proteingebundenes Ca2+-CaM. Die Protein Data Bank (PDB) accession numbers für die drei Proteine sind 1CFC (Apo-CaM), 1CLL (Ca2+-CaM) und 1CFF (proteingebundenes Ca2+-CaM)
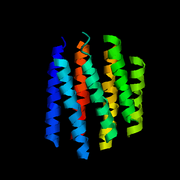
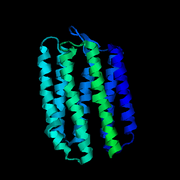
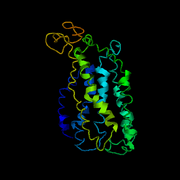
Web-Abb. 2.8
Strukturen verschiedener heptahelicaler (7-TMS) Rezeptoren. (A) Der Photorezeptor Bacteriorhodopsin aus Halobacterium halobium. (B) Die A-Kette des Photorezeptor Sensory Rhodopsin II aus Natronomonas pharaonis. (C) Die A-Kette dunkeladaptierten Rinder-Rhodopsins. Bei allen drei Molekülen sind die charakteristischen sieben Transmembran-Domänen erkennbar. Die Protein Data Bank (PDB) accession numbers für die drei Proteine sind 1BRD (Bacteriorhodopsin), 1GU8 (Sensory Rhodopsin II, Chain A) und 1JFP (Bovine Rhodopsin, Chain A).
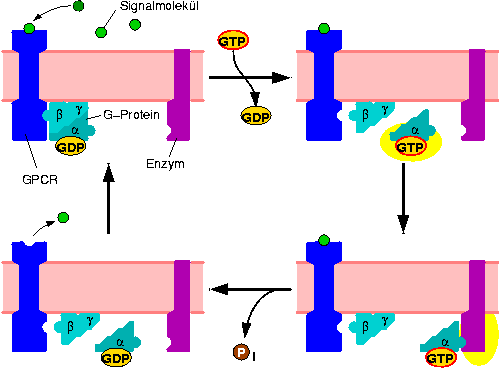
Web-Abb. 2.9
Der Zyklus der heterotrimeren G-Proteine bei Säugetieren - Vorbild auch für Pflanzen? Aktivierung des Rezeptor durch Bindung eines Liganden aktiviert das G-Protein durch den induzierten Austausch von GDP durch GTP an der α-Untereinheit des G-Proteins. Daraufhin dissoziiert das G-Protein vom Rezeptor und Gα von Gβγ. Gα und/oder Gβγ können nun mit Effektor-Proteinen in Wechselwirkung treten. Eine endogene GTPase-Aktivität von Gα spaltet Pi vom gebundenen GTP, inaktiviert damit Gα und sorgt für die Reassoziation des Trimers. Nach [Buchanan, 2000Buchanan, B.; Gruissem, W.; Jones, R.L. (Hg.) (2000): Biochemistry and Molecular Biology of Plants, American Society of Plant Physiologists, Rockville], verändert.
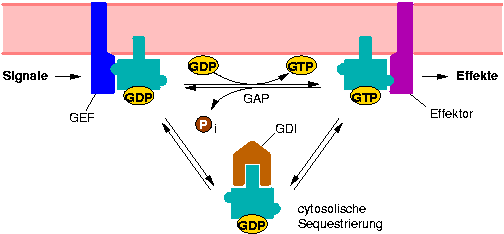
Web-Abb. 2.10
Der Zyklus der monomeren G-Proteine. Ein Signal stimuliert den Guaninnukleotid-Austauschfaktor (guanine nucleotide exchange factor, GEF), der daraufhin das monomere G-Protein durch GDP/GTP-Austausch aktiviert. Das GTP-gebundene aktive G-Protein interagiert mit seiner Effektordomäne mit einem oder mehreren spezifischen nachgeschalteten Effektorproteinen. Das an der aktiven Form gebundene GTP wird durch Bindung eines GTPase-aktivierenden Proteins (GTPase-activating protein, GAP) zum GDP hydrolysiert und das G-Protein inaktiviert. Die Sequestrierung (Ablösung von der Membran) der GTPase durch den guanine nucleotide dissociation inhibitor (GDI) reguliert die GTPasen negativ, da nur membrangebundene monomere G-Proteine durch den GEF aktiviert werden können. Nach [Yang, 2002Yang, Zhenbiao (2002): Small GTPases: Versatile Signaling Switches in Plants, The Plant Cell 14:S375-388], verändert.
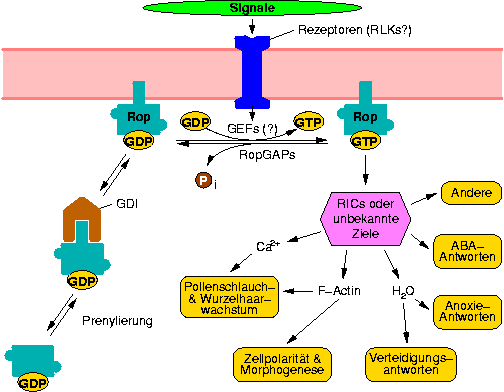
Web-Abb. 2.11
Funktionale Diversität des monomeren G-Proteins Rop als Signalmolekül in Pflanzen. Der Signalweg über Rop kontrolliert viele Prozesse und schließt viele mit Rop interagierende Proteine mit ein. Verschiedene Rops interagieren eventuell verschiedenen mit verschiedenenen Rop-Regulatoren und Zielproteinen: Dadurch entstehen funktional verschiedene Rop-Signalwege. Die Regulation von Rop erfolgt nicht nur durch Aktivierung (unbekannter Mechanismus) und Deaktivierung durch RopGAPs und Rop GDIs, sondern auch durch Kompartimentierung in spezifischen Membranen oder Membrandomänen. Abkürzungen: GEF - guanine nucleotide exchange factor; GAP - GTPase-activating protein; GDI - guanine nucleotide dissociation inhibitor; RIC - ROP-interacting CRIB-containing protein; RLK - receptor-like kinase; ABA - abscisic acid. Nach [Yang, 2002Yang, Zhenbiao (2002): Small GTPases: Versatile Signaling Switches in Plants, The Plant Cell 14:S375-388], verändert.
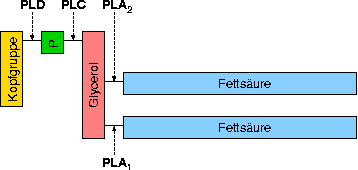
Web-Abb. 2.12
Spezifische Schnittstellen der verschiedenen Gruppen von Phospholipasen am Glycerophospholipid-Molekül. Die hydrophobe Komponente des Moleküls ist Diacylglycerol (DAG), das aus einem Glycerolrückgrad besteht, an das zwei langkettige Fettsäuren durch Esterbindungen gebunden sind. Dieser Teil des Moleküls ist durch eine Phosphodiester-Bindung an verschiedene polare Kopfgruppen gebunden, die die verschiedenen Klassen von Phospholipiden definieren. Eingezeichnet sind die spezifischen Spaltungsstellen für die Phospholipasen A1, A2, C und D. Nach [Munnik, 1998Munnik, T.; Irvine, R.F.; Musgrave, A. (1998): Phospholipid signalling in plants, Biochimica et Biophysica Acta 1389:222-272], verändert.
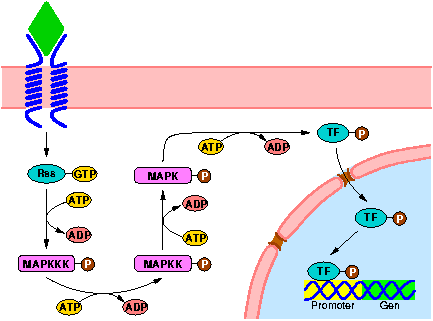
Web-Abb. 2.13
Die MAPK-Kaskade als zentrales Signaltransduktions-Modul bei Tieren: ein brauchbares Modell für Pflanzen? Ein Rezeptor aktiviert (über heterotrimere G-Proteine) das monomere G-Protein Ras, das daraufhin die MAPK Kinase Kinase (MAPKKK) phosphoryliert. Die MAPKKK phosphoryliert ihrerseits die MAPK Kinase (MAPKK), diese die MAP-Kinase (MAPK). Die MAPK phosphoryliert schließlich einen Transkriptionsfaktor (TF), der in den Zellkern einwandert, und dort durch Bindung an den Promoter eines spezifischen Gens dessen Expression reguliert. Nach [Buchanan, 2000Buchanan, B.; Gruissem, W.; Jones, R.L. (Hg.) (2000): Biochemistry and Molecular Biology of Plants, American Society of Plant Physiologists, Rockville], verändert