Inhaltsverzeichnis
Flavoproteine
Flavine
Flavine sind die am weitesten verbreiteten organischen Kofaktoren von Proteinen [Reid, 2002Reid, G. A. (2002): Flavins, Flavoproteins and Flavoproteomics, in Chapman, S.; Perham, R.; Scrutton, N. (Hg.): Flavins and Flavoproteins 2002, S. 3, Rudolf Weber, Berlin, Ames, 2002Ames, Bruce N.; Elson-Schwab, Ilan; Silver, Eli A. (2002): High-dose vitamin therapy stimulates variant enzymes with decreased coenzyme binding affinity (increased Km): relevance to genetic disease and polymorphisms, Am. J. Clin. Nutr. 75:616-658, Massey, 1995Massey, Vincent (1995): Introduction: flavoprotein structure and mechanism, FASEB J. 9:473-475, Massey, 2000Massey, Vincent (2000): The Chemical and Biological Versatility of Riboflavin, Biochem. Soc. Trans. 28:283-296]. Grund dafür ist die Vielfalt ihrer möglichen Redoxzustände: In biologischer Umgebung können sie voll reduziert, ein-Elektron-reduziert (Semichinon, Radikal) und voll oxidiert vorkommen. Das erlaubt ihnen, an einer Vielzahl biochemischer Reaktionen als Redoxkatalysatoren in Ein- und Zwei-Elektronen-Transferreaktionen teilzunehmen und insbesondere zwischen der Zwei-Elektronen-Oxidation der meisten organischen Substrate und den Ein–Elektronen-Reaktionen der Atmungskette zu vermitteln [Massey, 2000Massey, Vincent (2000): The Chemical and Biological Versatility of Riboflavin, Biochem. Soc. Trans. 28:283-296]. Darüber hinaus agieren sie als Elektrophile oder Nukleophile und bilden häufig während der Katalyse kovalente Intermediate mit dem Substrat, aber auch mit dem Protein [Mewies, 1998Mewies, Martin; McIntire, William S.; Scrutton, Nigel S. (1998): Covalent attachment of flavin adenine dinucleotide (FAD) and flavin mononucleotide (FMN) to enzymes: The current state of affairs, Protein Sci. 7:7, Edmondson, 2001Edmondson, Dale E.; Newton-Vinson, Paige (2001): The Covalent FAD of Monoamine Oxidase: Structural and Functional Role and Mechanism of the Flavinylation Reaction, Antioxid. Redox Signal. 3:789-806].
Jeder der drei Redoxzustände des Flavins hat ein charakteristisches UV/Vis–Spektrum (vgl. Abb. 1). Das erlaubt, mit Hilfe der optischen Spektroskopie den Redoxzustand des Flavins im Protein sicher zu bestimmen. Gleichzeitig kann anhand der Form charakteristischer Absorptionsbanden zwischen freiem und proteingebundenem Flavin unterschieden werden.
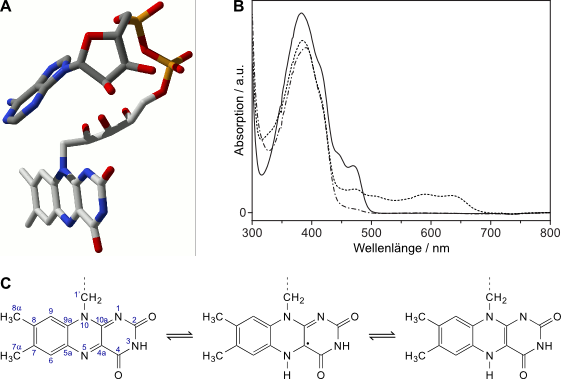
Abbildung 1: Der Flavinkofaktor der Photolyasen und Cryptochrome. (A): U-förmige Konformation des Flavinadenindinukleotids (FAD), wie sie in allen Photolyasen und Cryptochromen konserviert ist, hier aus Synechocystis sp. Cry-DASH, PDB ID 1NP7. (B, C): Die drei biologisch relevanten Redoxzustände von Flavin: (B) UV/Vis-Spektren der drei Redoxzustände von Flavin (proteingebunden, Xenopus laevis Cryptochrom-DASH): FADox (-), FADrad (- -) und FADred (-.-). Das Absorptionsmaximum bei 380 nm stammt vom zweiten Kofaktor des Proteins (MTHF). (C) Strukturformeln des 7,8-Dimethylisoalloxazinrestes des Flavins in seinen drei Oxidationszuständen: FADox (links), FADrad (mittig) und FADred (rechts). In FADox ist die Nummerierung der Atome nach IUPAC eingezeichnet. (A) wurde mit dem Programm spdbv [Guex, 1997Guex, N.; Peitsch, M.C. (1997): SWISS-MODEL and the Swiss-PdbViewer: An environment for comparative protein modeling, Electrophoresis 18:2714-2723] erstellt.
Jeder der Redox-Zustände existiert in einer kationischen, neutralen und anionischen Form, abhängig vom pH-Wert der Umgebung. Der voll oxidierte Redox-Zustand ist typischerweise der energetisch stabilste in Flavoproteinen.
Flavine absorbieren Licht im sichtbaren Wellenlängenbereich: Die optische Absorption des Grundzustands der Flavine liegt zwischen 400 und 500 nm. Zusammen mit der hohen Quantenausbeute für das Intersystem Crossing (ISC) und das hohe Oxidationspotential des Triplett-Zustandes ist diese Eigenschaft für die Photooxidation der Flavine verantwortlich.
Diese Eigenschaften sind ebenfalls Voraussetzung für die quantitative Generierung des Semichinon-Radikals und der Hydrochinon-Form der Flavine in Proteinen ausgehend vom voll oxidierten Zustand. Diese Photoreduktions-Reaktionen laufen typischerweise unter Blaulicht ab. Sie konnten auch ohne externe Elektronendonatoren in Proteinen, z.B. der DNA-Photolyase, beobachtet werden.
Blaulichtaktive Flavoproteine
Innerhalb der großen Gruppe der Flavoproteine gibt es drei Proteinfamilien, die die Absorption des Flavinkofaktors im blauen Bereich des sichtbaren Lichtes für ihre physiologische Funktion nutzen: Phototropine (allgemeiner: Proteine mit LOV–Domänen), BLUF-Proteine und Photolyasen/Cryptochrome [Losi, 2007Losi, Aba (2007): Flavin-based Blue-light Photosensors: A Photobiophysics Update, Photochem. Photobiol. 83:1283-1300]. Die Phototropine sind, wie der Name sagt, am lichtgerichteten Pflanzenwachstum (Phototropismus), aber auch der Chloroplastenbewegung, der Stomata–Öffnung in Schließzellen der Blätter, der schnellen Inhibition des Sproßwachstums und der Gametogenese (sexuelle Differenzierung in Algen) beteiligt. Die das Flavin tragende Proteindomäne wird als LOV-Domäne (Light Oxygen Voltage}, [Huala, 1997Huala, Eva; Oeller, Paul W.; Liscum, Emmanuel; Han, In-Seob; Larsen, Elise; Briggs, Winslow R. (1997): \emphArabidopsis NPH1: A Protein Kinase with a Putative Redox-Sensing Domain, Science 278:2120-2123]) bezeichnet. Proteine mit LOV–Domänen kommen neben in Pflanzen auch in Bakterien vor [Crosson, 2003Crosson, Sean; Rajagopal, Sudarshan; Moffat, Keith (2003): The LOV Domain Family: Photoresponsive Signaling Modules Coupled to Diverse Output Domains, Biochemistry 42:2-10, Losi, 2004Losi, Aba (2004): The bacterial counterparts of plant phototropins, Photochem. Photobiol. Sci. 3:566-574, Briggs, 2006Briggs, Winslow R. (2006): Flavin-Based Photoreceptors in Plants, in Silva, Eduardo; Edwards, Ana M. (Hg.): Flavins. Photochemistry and Photobiology, Kap. 9, S. 183-216, RSC Publishing, Cambridge, UK, Losi, 2006Losi, Aba (2006): Flavin-Based Photoreceptors in Bacteria, in Silva, Eduardo; Edwards, Ana M. (Hg.): Flavins. Photochemistry and Photobiology, Kap. 10, S. 217-269, RSC Publishing, Cambridge, UK, Losi, 2007Losi, Aba (2007): Flavin-based Blue-light Photosensors: A Photobiophysics Update, Photochem. Photobiol. 83:1283-1300]. Die BLUF–Proteine (Blue Light Using Flavin) agieren als redox- und lichtregulierte Derepressoren in Purpurbakterien und als Photorezeptoren für die Steuerung von Verhaltensreaktionen in Flagellaten wie Euglena [Gomelsky, 1995Gomelsky, Mark; Kaplan, Samuel (1995): \emphappA, a novel gene encoding a \emphtrans-acting factor involved in the regulation of photosynthesis gene expression in \emphRhodobacter sphaeroides 2.4.1, J. Bacteriol. 177:4609-4618, Gomelsky, 1998Gomelsky, Mark; Kaplan, Samuel (1998): AppA, a Redox Regulator of Photosystem Formation in \emphRhodobacter sphaeroides 2.4.1, Is a Flavoprotein. Identification of a Novel FAD Binding Domain, J. Biol. Chem. 273:35319-35325, Briggs, 2006Briggs, Winslow R. (2006): Flavin-Based Photoreceptors in Plants, in Silva, Eduardo; Edwards, Ana M. (Hg.): Flavins. Photochemistry and Photobiology, Kap. 9, S. 183-216, RSC Publishing, Cambridge, UK, Losi, 2006Losi, Aba (2006): Flavin-Based Photoreceptors in Bacteria, in Silva, Eduardo; Edwards, Ana M. (Hg.): Flavins. Photochemistry and Photobiology, Kap. 10, S. 217-269, RSC Publishing, Cambridge, UK].
Die Photolyase–Cryptochrom–Proteinfamilie besteht aus einer Reihe untereinander stark homologer Proteine: Photolyasen, die UV-Schäden an der DNA reparieren und ihrerseits nach zu reparierendem Photoprodukt in CPD- und (6-4) Photolyasen unterteilt werden [Sancar, 2003Sancar, Aziz (2003): Structure and Function of DNA Photolyase and Cryptochrome Blue-Light Photoreceptors, Chem. Rev. 103:2203-2237], Cryptochrome als Blaulichtrezeptoren, die sowohl im Pflanzen- als auch im Tierreich verbreitet und an der Regulation des zirkadianen Rhythmus beteiligt sind [Lin, 2005Lin, Chentao; Todo, Takeshi (2005): The cryptochromes, Genome Biology 6:220], und die erst vor relativ kurzer Zeit entdeckten DASH-Cryptochrome [Brudler, 2003Brudler, Ronald; Hitomi, Kenichi; Daiyasu, Hiromi; Toh, Hiroyuki; Kucho, Ken-ichi; Ishiura, Massahiro; Kaneshisa, Minoru; Roberts, Victoria A.; Todo, Takeshi; Tainer, John A.; Getzoff, Elisabeth D. (2003): Identification of a New Cryptochrome Class: Structure, Function, and Evolution, Mol. Cell 11:59-67]. Letztere sind strukturell den Photolyasen am ähnlichsten, zeigen aber keine (bzw. nur geringe) DNA–Reparaturaktivität. Ihre Funktion ist bisher für viele Vertreter noch nicht bekannt, offensichtlich aber stark heterogen.
Die Proteine werden hauptsächlich mit unterschiedlichen Methoden der EPR untersucht.
Literatur
- Ames, Bruce N.; Elson-Schwab, Ilan; Silver, Eli A. (2002): High-dose vitamin therapy stimulates variant enzymes with decreased coenzyme binding affinity (increased Km): relevance to genetic disease and polymorphisms, Am. J. Clin. Nutr. 75:616-658
- Briggs, Winslow R. (2006): Flavin-Based Photoreceptors in Plants, in Silva, Eduardo; Edwards, Ana M. (Hg.): Flavins. Photochemistry and Photobiology, Kap. 9, S. 183-216, RSC Publishing, Cambridge, UK
- Brudler, Ronald; Hitomi, Kenichi; Daiyasu, Hiromi; Toh, Hiroyuki; Kucho, Ken-ichi; Ishiura, Massahiro; Kaneshisa, Minoru; Roberts, Victoria A.; Todo, Takeshi; Tainer, John A.; Getzoff, Elisabeth D. (2003): Identification of a New Cryptochrome Class: Structure, Function, and Evolution, Mol. Cell 11:59-67
- Crosson, Sean; Rajagopal, Sudarshan; Moffat, Keith (2003): The LOV Domain Family: Photoresponsive Signaling Modules Coupled to Diverse Output Domains, Biochemistry 42:2-10
- Edmondson, Dale E.; Newton-Vinson, Paige (2001): The Covalent FAD of Monoamine Oxidase: Structural and Functional Role and Mechanism of the Flavinylation Reaction, Antioxid. Redox Signal. 3:789-806
- Gomelsky, Mark; Kaplan, Samuel (1995): \emphappA, a novel gene encoding a \emphtrans-acting factor involved in the regulation of photosynthesis gene expression in \emphRhodobacter sphaeroides 2.4.1, J. Bacteriol. 177:4609-4618
- Gomelsky, Mark; Kaplan, Samuel (1998): AppA, a Redox Regulator of Photosystem Formation in \emphRhodobacter sphaeroides 2.4.1, Is a Flavoprotein. Identification of a Novel FAD Binding Domain, J. Biol. Chem. 273:35319-35325
- Guex, N.; Peitsch, M.C. (1997): SWISS-MODEL and the Swiss-PdbViewer: An environment for comparative protein modeling, Electrophoresis 18:2714-2723
- Huala, Eva; Oeller, Paul W.; Liscum, Emmanuel; Han, In-Seob; Larsen, Elise; Briggs, Winslow R. (1997): \emphArabidopsis NPH1: A Protein Kinase with a Putative Redox-Sensing Domain, Science 278:2120-2123
- Lin, Chentao; Todo, Takeshi (2005): The cryptochromes, Genome Biology 6:220
- Losi, Aba (2004): The bacterial counterparts of plant phototropins, Photochem. Photobiol. Sci. 3:566-574
- Losi, Aba (2006): Flavin-Based Photoreceptors in Bacteria, in Silva, Eduardo; Edwards, Ana M. (Hg.): Flavins. Photochemistry and Photobiology, Kap. 10, S. 217-269, RSC Publishing, Cambridge, UK
- Losi, Aba (2007): Flavin-based Blue-light Photosensors: A Photobiophysics Update, Photochem. Photobiol. 83:1283-1300
- Massey, Vincent (1995): Introduction: flavoprotein structure and mechanism, FASEB J. 9:473-475
- Massey, Vincent (2000): The Chemical and Biological Versatility of Riboflavin, Biochem. Soc. Trans. 28:283-296
- Mewies, Martin; McIntire, William S.; Scrutton, Nigel S. (1998): Covalent attachment of flavin adenine dinucleotide (FAD) and flavin mononucleotide (FMN) to enzymes: The current state of affairs, Protein Sci. 7:7
- Reid, G. A. (2002): Flavins, Flavoproteins and Flavoproteomics, in Chapman, S.; Perham, R.; Scrutton, N. (Hg.): Flavins and Flavoproteins 2002, S. 3, Rudolf Weber, Berlin
- Sancar, Aziz (2003): Structure and Function of DNA Photolyase and Cryptochrome Blue-Light Photoreceptors, Chem. Rev. 103:2203-2237
